Drosten ist ein Wissenschaftsdarsteller. Das hat er bereits bei bei der Schweinegrippe bewiesen und beweist es jetzt erneut. Der sogenannte Test ist reiner Hokuspokus. Seine Veröffenlichung ist grob Fehlerhaft, und, gelinde gesagt, durch Korruption begleitet.
Das englische Original:
https://cormandrostenreview.com/report/
Hier die deutsche Übersetzung:
Übersichtsbericht Corman-Drosten et al. Eurosurveillance 2020
Dieser umfangreiche Review-Bericht wurde offiziell am 27. November 2020 über das Einreichungsportal der Eurosurveillance-Redaktion eingereicht. Diesem Review-Bericht ist ein Retraction Request Letter beigefügt, der von allen Haupt- und Co-Autoren unterzeichnet ist. Der erste und letzte aufgelistete Name sind der erste und zweite Hauptautor. Alle Namen dazwischen sind Co-Autoren.
Externes Peer-Review des RTPCR-Tests zum Nachweis von SARS-CoV-2 zeigt 10 wesentliche wissenschaftliche Fehler auf molekularer und methodischer Ebene: Konsequenzen für falsch positive Ergebnisse.
Pieter Borger(1), Bobby Rajesh Malhotra(2) , Michael Yeadon(3) , Clare Craig(4), Kevin McKernan(5) , Klaus Steger(6) , Paul McSheehy(7) , Lidiya Angelova(8), Fabio Franchi(9), Thomas Binder(10), Henrik Ullrich(11) , Makoto Ohashi(12), Stefano Scoglio(13), Marjolein Doesburg-van Kleffens(14), Dorothea Gilbert(15), Rainer Klement(16), Ruth Schruefer(17), Berber W. Pieksma(18), Jan Bonte(19), Bruno H. Dalle Carbonare(20), Kevin P. Corbett(21), Ulrike Kämmerer(22)
ABSTRACT
In der Publikation mit dem Titel „Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR“ (Eurosurveillance 25(8) 2020) stellen die Autoren einen diagnostischen Arbeitsablauf und ein RT-qPCR-Protokoll für den Nachweis und die Diagnostik von 2019-nCoV (jetzt bekannt als SARS-CoV-2) vor, von dem sie behaupten, dass er validiert ist und eine robuste diagnostische Methode für den Einsatz in Public-Health-Labors darstellt.
In Anbetracht der Konsequenzen, die sich aus dieser Publikation für Gesellschaften weltweit ergeben, hat eine Gruppe unabhängiger Forscher eine Punkt-für-Punkt-Überprüfung der oben genannten Publikation durchgeführt, bei der 1) alle Komponenten des vorgestellten Testdesigns gegengeprüft wurden, 2) die RT-qPCR-Protokoll-Empfehlungen im Hinblick auf die gute Laborpraxis bewertet wurden und 3) die Parameter anhand der relevanten wissenschaftlichen Literatur auf diesem Gebiet untersucht wurden.
Das veröffentlichte RT-qPCR-Protokoll zum Nachweis und zur Diagnostik von 2019-nCoV und das Manuskript weisen zahlreiche technische und wissenschaftliche Fehler auf, darunter ein unzureichendes Primerdesign, ein problematisches und unzureichendes RT-qPCR-Protokoll und das Fehlen einer genauen Testvalidierung. Weder der vorgestellte Test noch das Manuskript selbst erfüllen die Anforderungen an eine akzeptable wissenschaftliche Publikation. Weiterhin werden gravierende Interessenkonflikte der Autoren nicht erwähnt. Schließlich deutet die sehr kurze Zeitspanne zwischen Einreichung und Annahme der Publikation (24 Stunden) darauf hin, dass ein systematischer Peer-Review-Prozess hier entweder nicht durchgeführt wurde oder von problematisch schlechter Qualität ist. Wir liefern überzeugende Belege für mehrere wissenschaftliche Unzulänglichkeiten, Fehler und Mängel.
In Anbetracht der hier dargestellten wissenschaftlichen und methodischen Mängel sind wir zuversichtlich, dass die Redaktion von Eurosurveillance keine andere Wahl hat, als die Publikation zurückzuziehen.
KURZER ÜBERSICHTSBERICHT
Dieses Papier wird zahlreiche schwerwiegende Fehler im Corman-Drosten-Papier aufzeigen, deren Bedeutung zu weltweiten Fehldiagnosen von Infektionen geführt hat, die SARS-CoV-2 zugeschrieben werden und mit der Krankheit COVID-19 in Verbindung stehen. Wir sind mit strengen Abriegelungen konfrontiert, die das Leben und die Lebensgrundlage vieler Menschen zerstört haben, den Zugang zu Bildung einschränken und diese von Regierungen auf der ganzen Welt auferlegten Beschränkungen sind ein direkter Angriff auf die Grundrechte der Menschen und ihre persönlichen Freiheiten, was zu Kollateralschäden für ganze Volkswirtschaften im globalen Maßstab führt.
Es gibt zehn fatale Probleme mit dem Corman-Drosten-Papier, die wir in den folgenden Abschnitten skizzieren und näher erläutern werden.
Das erste und größte Problem ist, dass das neuartige Coronavirus SARS-CoV-2 (in der Publikation als 2019-nCoV und im Februar 2020 von einem internationalen Konsortium von Virusexperten als SARS-CoV-2 bezeichnet) auf in silico (theoretischen) Sequenzen basiert, die von einem Labor in China geliefert wurden [1], weil den Autoren zum damaligen Zeitpunkt weder Kontrollmaterial von infektiösem („live“) oder inaktiviertem SARS-CoV-2 noch isolierte genomische RNA des Virus zur Verfügung standen. Bis heute wurde von der Autorenschaft keine Validierung auf der Basis von isolierten SARS-CoV-2-Viren oder deren Volllängen-RNA durchgeführt. Laut Corman et al:
„Unser Ziel war es, eine robuste Diagnosemethode für den Einsatz in Labors des öffentlichen Gesundheitswesens zu entwickeln und einzusetzen, ohne dass Virusmaterial zur Verfügung stand.“ [1]
Der Fokus sollte hier auf die beiden genannten Ziele gelegt werden: a) Entwicklung und b) Einsatz eines diagnostischen Tests für den Einsatz in öffentlichen Gesundheitslabors. Diese Ziele sind ohne die Verfügbarkeit von tatsächlichem Virusmaterial (z. B. zur Bestimmung der infektiösen Viruslast) nicht zu erreichen. In jedem Fall kann nur ein Protokoll mit maximaler Genauigkeit das zwingende und primäre Ziel in einem Szenario dieser Größenordnung sein. Die Bestimmung der kritischen Viruslast ist eine Pflichtinformation, und es liegt in der Verantwortung der Gruppe von Christian Drosten, diese Experimente durchzuführen und die entscheidenden Daten zu liefern.
Nichtsdestotrotz wurden diese in silico-Sequenzen verwendet, um eine RT-PCR-Testmethodik zur Identifizierung des besagten Virus zu entwickeln. Dieses Modell basierte auf der Annahme, dass das neue Virus dem SARS-CoV aus dem Jahr 2003 sehr ähnlich ist, da es sich bei beiden um Beta-Coronaviren handelt.
Der PCR-Test wurde daher unter Verwendung der genomischen Sequenz von SARS-CoV als Kontrollmaterial für die Sarbeco-Komponente entworfen; wir wissen dies aus unserer persönlichen E-Mail-Kommunikation mit [2], einem der Co-Autoren der Corman-Drosten-Arbeit. Diese Methode zur Modellierung von SARS-CoV-2 wurde im Corman-Drosten-Paper wie folgt beschrieben:
„die Etablierung und Validierung eines diagnostischen Arbeitsablaufs für das 2019-nCoV-Screening und die spezifische Bestätigung, der in Abwesenheit von verfügbaren Virusisolaten oder Original-Patientenproben entwickelt wurde. Design und Validierung wurden durch die enge genetische Verwandtschaft mit dem SARS-CoV von 2003 ermöglicht und durch den Einsatz der synthetischen Nukleinsäure-Technologie unterstützt.“
Die Reverse Transkriptions-Polymerase-Kettenreaktion (RT-PCR) ist eine wichtige biomolekulare Technologie zum schnellen Nachweis von seltenen RNA-Fragmenten, die im Voraus bekannt sind. Im ersten Schritt werden die in der Probe vorhandenen RNA-Moleküle revers transkribiert, um cDNA zu erhalten. Die cDNA wird dann in der Polymerase-Kettenreaktion unter Verwendung eines spezifischen Primerpaares und eines thermostabilen DNA-Polymerase-Enzyms amplifiziert. Die Technologie ist hochsensitiv und ihre Nachweisgrenze liegt theoretisch bei 1 Molekül cDNA. Die Spezifität der PCR wird stark durch biomolekulare Designfehler beeinflusst.
Was ist beim Design eines RT-PCR-Tests und des in der Corman-Drosten-Publikation beschriebenen quantitativen RT-qPCR-Tests wichtig?
- Die Primer und Sonden:
- Die Konzentration der Primer und Sonden muss im optimalen Bereich liegen
- (100-200 nM)
- müssen spezifisch für das zu amplifizierende Zielgen sein
- müssen einen optimalen prozentualen Anteil an GC-Gehalt im Verhältnis zu den gesamten stickstoffhaltigen Basen haben (mindestens 40%, maximal 60%)
- für die Virusdiagnostik müssen mindestens 3 Primerpaare 3 virale Gene detektieren (vorzugsweise möglichst weit voneinander entfernt im viralen Genom)
- Die Temperatur, bei der alle Reaktionen ablaufen:
- DNA-Schmelztemperatur (>92°)
- DNA-Amplifikationstemperatur (TaqPol spezifisch)
- Tm; die Annealing-Temperatur (die Temperatur, bei der die Primer und Sonden die Zielbindung/-ablösung erreichen, nicht mehr als 2 °C pro Primerpaar). Tm hängt stark vom GC-Gehalt der Primer ab
- Die Anzahl der Amplifikationszyklen (weniger als 35; vorzugsweise 25-30 Zyklen);
Im Falle des Virusnachweises werden bei >35 Zyklen nur Signale detektiert, die nicht mit infektiösen Viren korrelieren, wie sie durch Isolierung in Zellkulturen bestimmt werden [reviewed in 2]; wenn jemand durch PCR als positiv getestet wird, wenn ein Schwellenwert von 35 Zyklen oder höher verwendet wird (wie es in den meisten Labors in Europa & den USA der Fall ist), ist die Wahrscheinlichkeit, dass diese Person tatsächlich infiziert ist, weniger als 3%, die Wahrscheinlichkeit, dass dieses Ergebnis ein falsches Positiv ist, beträgt 97% [reviewed in 3] - Molekularbiologische Validierungen; amplifizierte PCR-Produkte müssen entweder durch den Lauf der Produkte in einem Gel mit einem DNA-Lineal oder durch direkte DNA-Sequenzierung validiert werden
- Positiv- und Negativkontrollen sollten angegeben werden, um den spezifischen Virusnachweis zu bestätigen/zu widerlegen
- Es sollte eine Standardarbeitsanweisung (Standard Operational Procedure, SOP) vorhanden sein
Die SOP legt die oben genannten Parameter eindeutig fest, so dass alle Labore in der Lage sind, die exakt gleichen Testbedingungen einzurichten. Eine validierte, allgemeingültige SOP ist unerlässlich, da sie den Vergleich von Daten innerhalb und zwischen Ländern ermöglicht.
KLEINERE BEDENKEN MIT DEM CORMAN-DROSTEN PAPIER
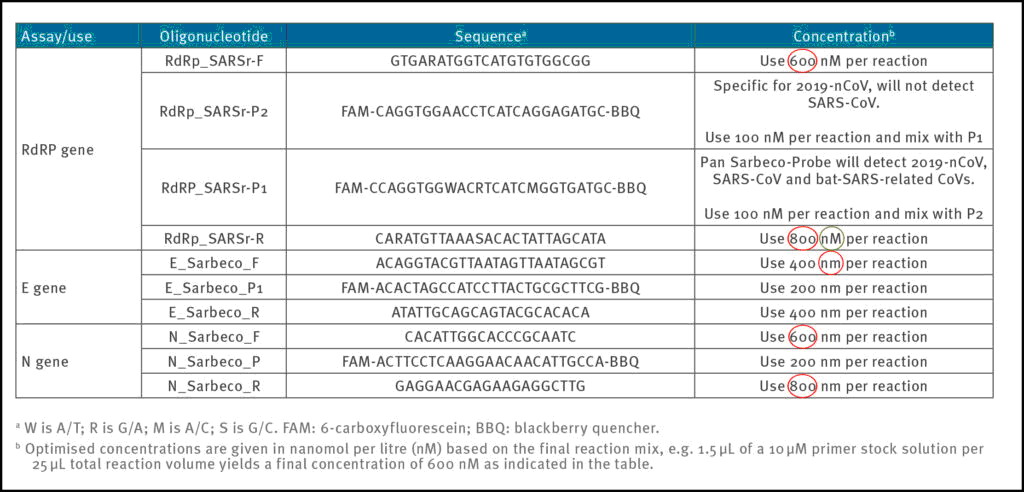
- In Tabelle 1 des Corman-Drosten-Papiers werden unterschiedliche Abkürzungen angegeben – „nM“ ist angegeben, „nm“ nicht. Weiter in Bezug auf die korrekte Nomenklatur: nm bedeutet „Nanometer“, daher sollte hier nM stehen.
- Es ist allgemeiner Konsens, genetische Sequenzen immer in 5′-3′-Richtung zu schreiben, auch die Reverse-Primer. Es ist höchst ungewöhnlich, ein Alignment mit rückwärts komplementärer Schreibweise der Primersequenz durchzuführen, wie es die Autoren in Abbildung 2 der Corman-Drosten-Arbeit getan haben. Hier wird zusätzlich eine Wobble-Base als „y“ markiert, ohne Beschreibung, für welche Basen das Y steht.
- Zwei irreführende Fallstricke in der Corman-Drosten-Arbeit sind, dass ihre Tabelle 1 weder Tm-Werte (Annealing-Temperatur-Werte) noch GC-Werte (Anzahl von G und C in den Sequenzen als %-Wert der Gesamtbasen) enthält.
HAUPTBEDENKEN GEGEN DAS CORMAN-DROSTEN-PAPIER
A) HINTERGRUND
Die Autoren stellen den Hintergrund für ihre wissenschaftliche Arbeit wie folgt vor: „Der anhaltende Ausbruch des kürzlich aufgetauchten neuartigen Coronavirus (2019-nCoV) stellt eine Herausforderung für die Labore des öffentlichen Gesundheitswesens dar, da Virusisolate nicht verfügbar sind, während sich die Hinweise häufen, dass der Ausbruch weiter verbreitet ist als ursprünglich angenommen und eine internationale Verbreitung durch Reisende bereits stattfindet“.
Laut BBC News [4] und Google Statistics [5] gab es am 21. Januar 2020 – dem Tag, an dem das Manuskript eingereicht wurde – weltweit 6 Todesfälle. Warum gingen die Autoren von einer Herausforderung für die Labore des öffentlichen Gesundheitswesens aus, obwohl es zu diesem Zeitpunkt keine substanziellen Hinweise darauf gab, dass der Ausbruch weiter verbreitet war als zunächst angenommen?
Als Ziel erklärten die Autoren, eine robuste diagnostische Methodik für den Einsatz in Labors des öffentlichen Gesundheitswesens zu entwickeln und einzusetzen, ohne Virusmaterial zur Verfügung zu haben. Weiterhin räumen sie ein, dass „die vorliegende Studie die enorme Reaktionsfähigkeit demonstriert, die durch die Koordination von akademischen und öffentlichen Laboren in nationalen und europäischen Forschungsnetzwerken erreicht wurde.“
B) METHODEN UND ERGEBNISSE
1. Primer- und Sondendesign
1a) Falsche Primer-Konzentrationen
Zuverlässige und genaue PCR-Testprotokolle werden normalerweise mit einer Konzentration zwischen 100 nM und 200 nM pro Primer entworfen [7]. In der Arbeit von Corman-Drosten beobachten wir ungewöhnlich hohe und variierende Primerkonzentrationen für mehrere Primer (Tabelle 1). Für die Primerpaare RdRp_SARSr-F und RdRp_SARSr-R werden 600 nM bzw. 800 nM beschrieben. In ähnlicher Weise werden für die Primerpaare N_Sarbeco_F und N_Sarbeco_R 600 nM bzw. 800 nM angegeben [1].
Es sollte klar sein, dass diese Konzentrationen viel zu hoch sind, um für spezifische Amplifikationen von Zielgenen optimal zu sein. Es gibt keinen spezifizierten Grund, diese extrem hohen Konzentrationen von Primern in diesem Protokoll zu verwenden. Vielmehr führen diese Konzentrationen zu einer erhöhten unspezifischen Bindung und PCR-Produktamplifikation.
Tabelle 1: Primers and probes (adapted from Corman-Drosten paper; erroneous primer concentrations are highlighted)
1b) Unspezifizierte („Wobbly“) Primer- und Sondensequenzen
Um reproduzierbare und vergleichbare Ergebnisse zu erhalten, ist es unerlässlich, die Primerpaare eindeutig zu definieren. In der Arbeit von Corman-Drosten haben wir sechs unspezifizierte Positionen beobachtet, die mit den Buchstaben R, W, M und S gekennzeichnet sind (Tabelle 2). Der Buchstabe W bedeutet, dass an dieser Position entweder ein A oder ein T stehen kann; R bedeutet, dass entweder ein G oder ein A stehen kann; M bedeutet, dass die Position entweder ein A oder ein C sein kann; der Buchstabe S bedeutet, dass an dieser Position entweder ein G oder ein C stehen kann.
Diese hohe Anzahl von Varianten ist nicht nur ungewöhnlich, sondern auch sehr verwirrend für die Labore. Diese sechs nicht spezifizierten Positionen könnten leicht zum Design mehrerer verschiedener alternativer Primersequenzen führen, die sich nicht auf SARS-CoV-2 beziehen (2 verschiedene RdRp_SARSr_F-Primer + 8 verschiedene RdRp_SARS_P1-Sonden + 4 verschiedene RdRp_SARSr_R). Die Design-Variationen führen zwangsläufig zu Ergebnissen, die nicht einmal mit SARS CoV-2 in Verbindung gebracht werden können. Daher ist die verwirrende unspezifische Beschreibung im Corman-Drosten-Papier nicht als Standard-Operationsprotokoll geeignet. Diese unspezifizierten Positionen hätten eindeutig gestaltet werden müssen.
Diese wackeligen Sequenzen haben bereits für Unruhe im Feld gesorgt und zu einem Letter to the Editor von Pillonel et al. [8] über eklatante Fehler in den beschriebenen Sequenzen geführt. Diese Fehler sind auch in der Beilage von Corman et al. offenkundig.
Tabelle 2: Primers and probes (adapted from Corman-Drosten paper; unspecified (“Wobbly”) nucleotides in the primers are highlighted)
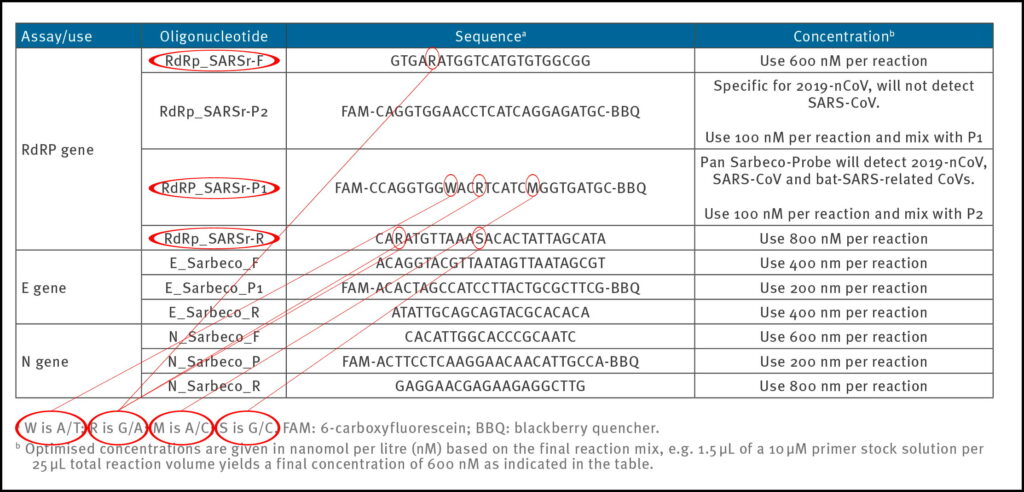
Das WHO-Protokoll (Abbildung 1), das sich direkt aus dem Corman-Drosten-Papier ableitet, kommt zu dem Schluss, dass zur Bestätigung des Vorhandenseins von SARS-CoV-2 zwei Kontrollgene (das E- und das RdRp-Gen) im Assay identifiziert werden müssen. Dabei ist zu beachten, dass das RdRd-Gen eine unsichere Position („wobbly“) im Vorwärts-Primer (R=G/A), zwei unsichere Positionen im Rückwärts-Primer (R=G/A; S=G/C) und drei unsichere Positionen in der RdRp-Sonde (W=A/T; R=G/A; M=A/C) hat. Es können also zwei verschiedene Vorwärtsprimer, vier verschiedene Rückwärtsprimer und acht verschiedene Sonden für das RdRd-Gen synthetisiert werden. Insgesamt gibt es 64 mögliche Kombinationen von Primern und Sonden!
Das Corman-Drosten-Papier identifiziert außerdem ein drittes Gen, das laut WHO-Protokoll nicht weiter validiert und als unnötig erachtet wurde:
„Bemerkenswert ist, dass der N-Gen-Assay ebenfalls gut abschnitt, aber nicht weiter intensiv validiert wurde, weil er etwas weniger empfindlich war.“
Dies war eine unglückliche Auslassung, da es am besten wäre, alle drei Gen-PCRs als Bestätigungsassays zu verwenden, und dies hätte zu einem fast ausreichenden Virus-RNA-Nachweis-Diagnosetool-Protokoll geführt. Drei konfirmatorische Assay-Schritte würden zumindest die Fehler und Unsicherheiten bei jedem Fold-Schritt in Bezug auf „Wobbly“-Spots minimieren. (Nichtsdestotrotz würde das Protokoll immer noch hinter jeder „guten Laborpraxis“ zurückbleiben, wenn man alle anderen Design-Fehler mit einbezieht).
So wie es aussieht, wird der N-Gentest bedauerlicherweise weder in der WHO-Empfehlung (Abbildung 1) als obligatorischer und entscheidender dritter Bestätigungsschritt vorgeschlagen, noch wird er im Corman-Drosten-Papier als wichtige optionale Absicherung „für einen Routine-Workflow“ hervorgehoben (Tabelle 2).
Infolgedessen wurden in fast allen Testverfahren weltweit lediglich 2 Primer-Matches statt aller drei verwendet. Dieses Versäumnis macht das gesamte Testprotokoll unbrauchbar, wenn es darum geht, exakte Testergebnisse zu liefern, die in einer laufenden Pandemie wirklich von Bedeutung sind.
Figure 1: Der N-Gene-Bestätigungs-Assay wird weder in der offiziellen WHO Drosten-Corman-Protokoll-Empfehlung unten [8] als notwendiger dritter Schritt hervorgehoben, noch wird er in der Euro-surveillance-Publikation als entscheidender Schritt für eine höhere Test-Genauigkeit gefordert.

1c) Falscher GC-Gehalt (diskutiert in 2c, zusammen mit Annealing-Temperatur (Tm))
1d) Nachweis von viralen Genen
Die RT-PCR wird für die Primärdiagnostik einer Infektion nicht empfohlen. Aus diesem Grund ist der in der klinischen Routine eingesetzte RT-PCR-Test zum Nachweis von COVID-19 nicht für die COVID-19-Diagnostik auf regulatorischer Basis indiziert.
„Kliniker müssen die verbesserte Genauigkeit und Geschwindigkeit der molekularen Diagnosetechniken für die Diagnose von Infektionen anerkennen, aber auch deren Grenzen verstehen. Laborergebnisse sollten immer im Zusammenhang mit der klinischen Präsentation des Patienten interpretiert werden, und für zuverlässige Testergebnisse sind der richtige Ort, die Qualität und der Zeitpunkt der Probenentnahme erforderlich.“ [9]
Er kann jedoch bei der Differentialdiagnose des Arztes helfen, wenn er zwischen verschiedenen Infektionen der Lunge unterscheiden muss (Grippe, Covid-19 und SARS haben sehr ähnliche Symptome). Für eine bestätigende Diagnose eines bestimmten Virus müssen mindestens 3 spezifische Primerpaare zum Nachweis von 3 virusspezifischen Genen eingesetzt werden. Vorzugsweise sollten diese Zielgene mit größtmöglichem Abstand im viralen Genom liegen (auch an den entgegengesetzten Enden).
In der Arbeit von Corman-Drosten werden zwar 3 Primer beschrieben, diese Primer decken aber nur etwa die Hälfte des Virusgenoms ab. Dies ist ein weiterer Faktor, der die Spezifität für den Nachweis von intakter COVID-19-Virus-RNA verringert und die Quote der falsch positiven Testergebnisse erhöht.
Selbst wenn wir also in einer Probe drei positive Signale erhalten (d. h. die drei Primerpaare ergeben drei verschiedene Amplifikationsprodukte), beweist dies nicht das Vorhandensein eines Virus. Ein besseres Primerdesign würde terminale Primer an beiden Enden des viralen Genoms haben. Dies liegt daran, dass das gesamte virale Genom abgedeckt wäre und drei positive Signale besser zwischen einem vollständigen (und damit potentiell infektiösen) Virus und fragmentierten viralen Genomen (ohne infektiöse Potenz) unterscheiden können. Um etwas Signifikantes über die Infektiosität des Virus abzuleiten, hätte das Orf1-Gen, das für das essentielle Replikase-Enzym der SARS-CoV-Viren kodiert, als Target aufgenommen werden müssen (Abbildung 2). Die Positionierung der Targets in der Region des viralen Genoms, die am stärksten und variabelsten transkribiert wird, ist eine weitere Schwäche des Protokolls.
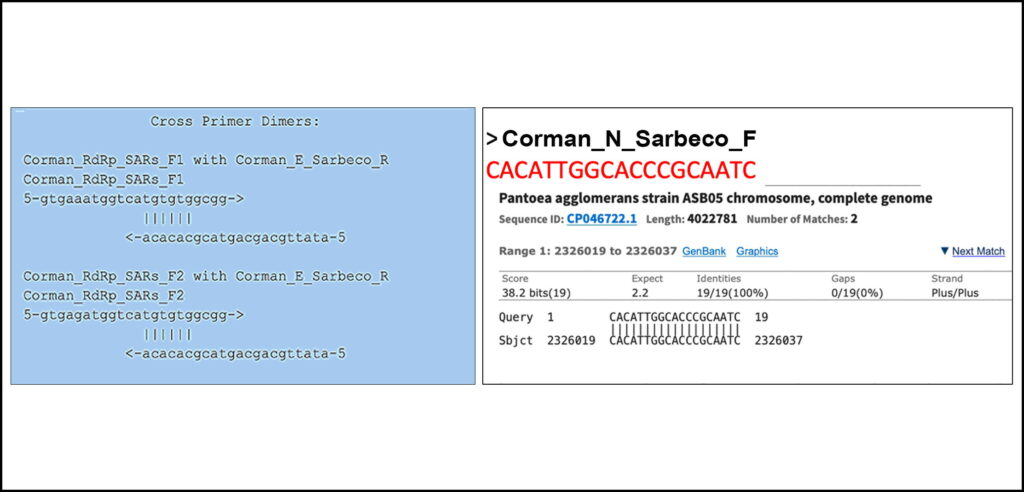
Kim et al. zeigen eine hochvariable 3′-Expression von subgenomischer RNA in Sars-CoV-2 [23]. Diese RNAs werden aktiv als Signaturen für asymptomatische und nicht-infektiöse Patienten überwacht [10]. Es ist höchst fragwürdig, eine Population von asymptomatischen Menschen mit qPCR-Primern zu screenen, die 6 Basenpaare Primer-Dimer am 3-Prime-Ende eines Primers haben (Abbildung 3).
Offenbar empfiehlt die WHO diese Primer. Wir testeten alle Wobble-Derivate aus der Corman-Drosten-Arbeit mit dem Primer-Dimer-Webtool von Thermofisher [11]. Der RdRp-Vorwärtsprimer hat 6bp 3-Prime-Homologie mit Sarbeco E Reverse. Bei hohen Primerkonzentrationen reicht dies aus, um Ungenauigkeiten zu erzeugen.
Bemerkenswert: Es gibt eine perfekte Übereinstimmung eines der N-Primer mit einem klinischen Erreger (Pantoea), der bei immungeschwächten Patienten gefunden wird. Der umgekehrte Primer trifft ebenfalls Pantoea, aber nicht in der gleichen Region (Abbildung 3).
Dies sind schwerwiegende Designfehler, da der Test nicht zwischen dem ganzen Virus und viralen Fragmenten unterscheiden kann. Der Test kann nicht als Diagnostikum für SARS-Viren verwendet werden.
Figure 2: Relative Positionen der Amplikonziele auf dem SARS-Coronavirus und dem neuen Coronavirus-Genom 2019. ORF: offener Leserahmen; RdRp: RNA-abhängige RNA-Polymerase. Die Zahlen unter dem Amplikon sind Genompositionen nach SARS-CoV, NC_004718 [1];
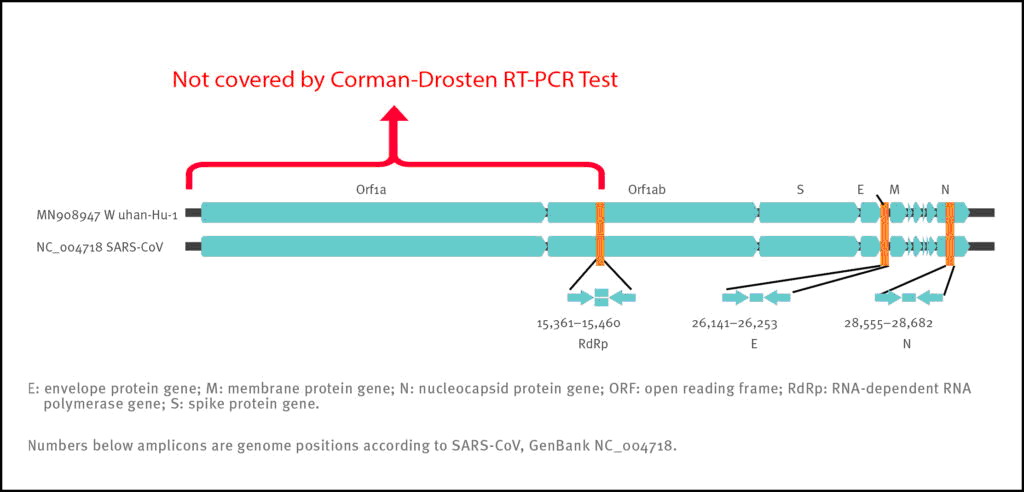 Figure 3: Abbildung 3: Ein Test mit dem Primer-Dimer-Webtool von Thermofischer zeigt, dass der RdRp-Vorwärtsprimer eine 6bp 3`Prime-Homologie mit Sarbeco E Reverse aufweist (linker Kasten). Ein weiterer Test zeigt, dass es eine perfekte Überein-stimmung für einen einen der N-Primer mit einem klinischen Erreger (Pantoea) gibt, der bei immungeschwächten Patienten gefunden wird (rechter Kasten).
Figure 3: Abbildung 3: Ein Test mit dem Primer-Dimer-Webtool von Thermofischer zeigt, dass der RdRp-Vorwärtsprimer eine 6bp 3`Prime-Homologie mit Sarbeco E Reverse aufweist (linker Kasten). Ein weiterer Test zeigt, dass es eine perfekte Überein-stimmung für einen einen der N-Primer mit einem klinischen Erreger (Pantoea) gibt, der bei immungeschwächten Patienten gefunden wird (rechter Kasten).
2. Reaktionstemperaturen
2a) DNA-Schmelztemperatur (>92°).
Wurde in der Arbeit von Corman-Drosten ausreichend behandelt.
2b) DNA-Amplifikationstemperatur.
In der Arbeit von Corman-Drosten ausreichend behandelt.
2c) Falsche GC-Gehalte und Tm
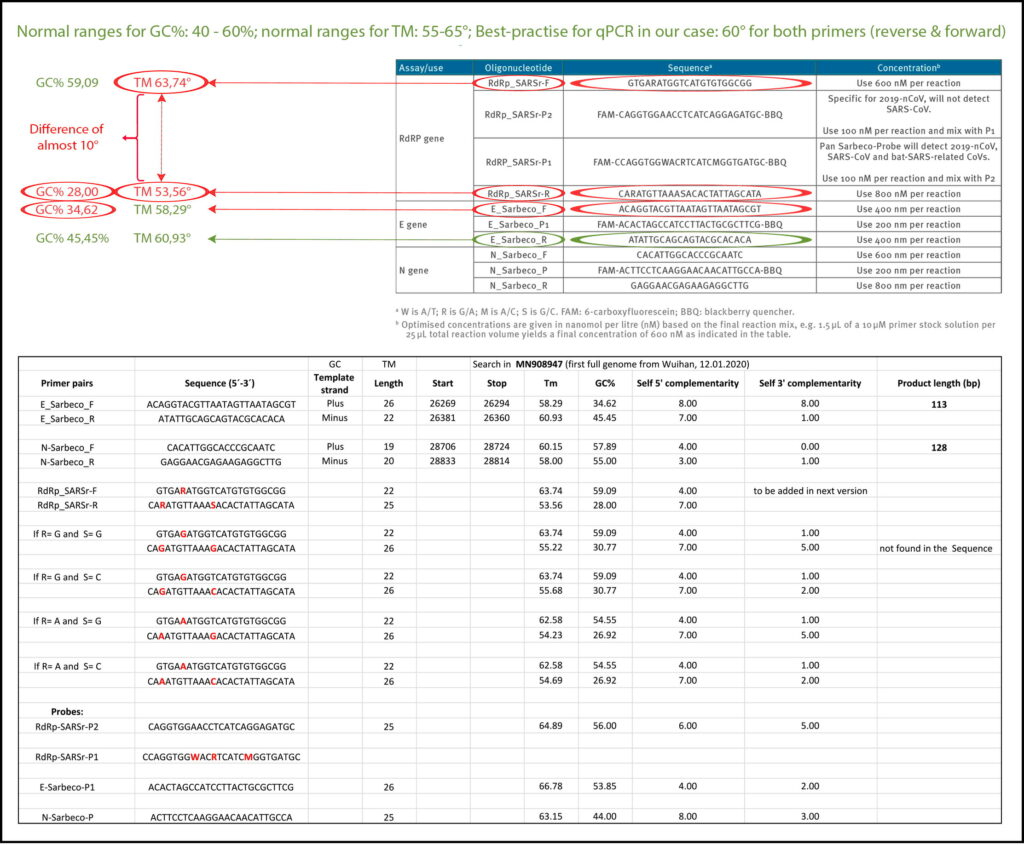
Die Annealing-Temperatur bestimmt, bei welcher Temperatur sich der Primer an die Zielsequenz anlagert bzw. von dieser ablöst. Für eine effiziente und spezifische Amplifikation sollte der GC-Gehalt der Primer mindestens 40 % und maximal 60 % der Amplifikation betragen. Wie in Tabelle 3 angegeben, liegen drei der in der Corman-Drosten-Arbeit beschriebenen Primer nicht im normalen Bereich für den GC-Gehalt. Zwei Primer (RdRp_SARSr_F und RdRp_SARSr_R) haben ungewöhnliche und sehr niedrige GC-Werte von 28 %-31 % für alle möglichen Varianten von Wobble-Basen, während der Primer E_Sarbeco_F einen GC-Wert von 34,6 % aufweist (Tabelle 3 und zweites Feld von Tabelle 3).
Es ist zu beachten, dass der GC-Gehalt aufgrund der drei Wasserstoffbrückenbindungen bei der Basenpaarung die Bindung an sein spezifisches Ziel weitgehend bestimmt. Je niedriger also der GC-Gehalt des Primers ist, desto geringer ist seine Bindungsfähigkeit an seine spezifische Zielgensequenz (d. h. das zu detektierende Gen). Das bedeutet, dass wir für die Erkennung einer Zielsequenz eine Temperatur wählen müssen, die möglichst nahe an der tatsächlichen Annealing-Temperatur (Best-Practice-Wert) liegt, damit sich der Primer nicht wieder ablöst, und gleichzeitig die Zielsequenz gezielt auswählen.
Wenn der Tm-Wert sehr niedrig ist, wie bei allen Wobbly-Varianten der RdRp-Reverse-Primer beobachtet, können die Primer unspezifisch an mehrere Targets binden, was die Spezifität verringert und potenziell falsch-positive Ergebnisse erhöht.
Die Annealing-Temperatur (Tm) ist ein entscheidender Faktor für die Bestimmung der Spezifität/Genauigkeit des qPCR-Verfahrens und wesentlich für die Bewertung der Genauigkeit von qPCR-Protokollen. Best-Practice-Empfehlung: Beide Primer (vorwärts und rückwärts) sollten einen annähernd gleichen Wert haben, vorzugsweise den gleichen Wert.
Wir benutzten die frei verfügbare Primer-Design-Software Primer-BLAST [12, 25], um die Best-Practice-Werte für alle in der Corman-Drosten-Arbeit verwendeten Primer auszuwerten (Tabelle 3). Wir versuchten, einen Tm-Wert von 60° C zu finden, während wir in ähnlicher Weise den höchstmöglichen GC%-Wert für alle Primer suchten. Eine maximale Tm-Differenz von 2° C innerhalb der Primerpaare wurde als akzeptabel angesehen. Beim Testen der in der Corman-Drosten-Arbeit angegebenen Primer-Paare haben wir für Primer-Paar1 (RdRp_SARSr_F und RdRp_SARSr_R) eine Differenz von 10° C in Bezug auf die Annealing-Temperatur Tm festgestellt. Dies ist ein sehr schwerer Fehler und macht das Protokoll als spezifisches Diagnosewerkzeug unbrauchbar.
Zusätzliche Tests zeigten, dass nur das Primerpaar, das für die Amplifikation des N-Gens entwickelt wurde (N_Sarbeco_F und N_Sarbeco_R), den adäquaten Standard erreicht, um in einem diagnostischen Test zu arbeiten, da es einen ausreichenden GC-Gehalt hat und die Tm-Differenz zwischen den Primern (N_Sarbeco_F und N_Sarbeco_R) 1,85° C beträgt (unterhalb des entscheidenden Maximums von 2° C Differenz). Wichtig ist, dass dies das Gen ist, das weder in den Virusproben getestet wurde (Tabelle 2) noch als Bestätigungstest hervorgehoben wurde. Zusätzlich zu den stark variierenden Schmelztemperaturen und degenerierten Sequenzen in diesen Primern gibt es einen weiteren Faktor, der die Spezifität des Verfahrens beeinflusst: Die dNTPs (0,4uM) sind 2x höher als für eine hochspezifische Amplifikation empfohlen. Außerdem wird der Reaktion zusätzlich Magnesiumsulfat zugesetzt. Dieses Verfahren in Kombination mit einer niedrigen Annealing-Temperatur kann zu unspezifischen Amplifikationen führen. Wenn zusätzliches Magnesium für die qPCR benötigt wird, sollte die Spezifität des Assays weiter hinterfragt werden.
Die hier beschriebenen Designfehler sind so schwerwiegend, dass eine spezifische Amplifikation von SARS-CoV-2-Genmaterial mit dem Protokoll der Corman-Drosten-Arbeit höchst unwahrscheinlich ist.
Tabelle 3: GC-Gehalt der Primer und Sonden (adaptiert aus Corman-Drosten paper; Abweichungen von optimierten GC-Gehalten sind hervorgehoben. Das zweite Panel zeigt eine tabellarische Auflistung aller Primer-BLAST Best-Practice-Werte für alle im Corman-Drosten-Paper verwendeten Primer und Sonden von Prof. Dr. Ulrike Kämmerer & ihrem Team
3. Die Anzahl der Amplifikationszyklen
Es ist anzumerken, dass im Corman-Drosten-Papier nirgends erwähnt wird, ob ein Test positiv oder negativ ist, oder was ein positives oder negatives Ergebnis definiert. Diese Arten von virologischen Diagnosetests müssen auf einer SOP basieren, einschließlich einer validierten und festgelegten Anzahl von PCR-Zyklen (Ct-Wert), nach denen eine Probe als positiv oder negativ eingestuft wird. Der maximale, einigermaßen zuverlässige Ct-Wert liegt bei 30 Zyklen. Oberhalb eines Ct-Wertes von 35 Zyklen muss mit einer schnell ansteigenden Anzahl von falsch-positiven Ergebnissen gerechnet werden.
PCR-Daten, die nach einem Ct-Wert von 35 Zyklen als positiv bewertet werden, sind völlig unzuverlässig.
Zitiert wird Jaafar et al. 2020 [3]: „Bei Ct = 35, dem Wert, den wir für die Meldung eines positiven PCR-Ergebnisses verwendet haben, sind <3 % der Kulturen positiv.“ Mit anderen Worten, es gab keine erfolgreiche Virusisolierung von SARS-CoV-2 bei diesen hohen Ct-Werten.
Weiterhin zeigen wissenschaftliche Studien, dass bei Ct-Werten von 35 nur nicht-infektiöse (tote) Viren nachgewiesen werden [22].
Zwischen 30 und 35 gibt es eine Grauzone, in der ein positiver Test nicht mit Sicherheit festgestellt werden kann. Dieser Bereich sollte ausgeschlossen werden. Natürlich könnte man 45 PCR-Zyklen durchführen, wie im Corman-Drosten WHO-Protokoll empfohlen (Abbildung 4), aber dann muss man auch einen vernünftigen Ct-Wert definieren (der 30 nicht überschreiten sollte). Ein Analysenergebnis mit einem Ct-Wert von 45 ist aber wissenschaftlich und diagnostisch absolut nicht aussagekräftig (ein vernünftiger Ct-Wert sollte nicht über 30 liegen). Dies alles sollte sehr deutlich kommuniziert werden. Es ist ein erheblicher Fehler, dass im Corman-Drosten-Papier der maximale Ct-Wert nicht erwähnt wird, bei dem eine Probe eindeutig als positives oder negatives Testergebnis gewertet werden kann. Dieser wichtige Zyklusgrenzwert wird auch in den bisherigen Nachfolgearbeiten nicht angegeben.
Figure 4: RT-PCR Kit Empfehlung im offiziellen Corman-Drosten WHO-Protokoll [8]. Es ist nur ein „Cycler“-Wert (Zyklen) zu finden ohne entsprechenden und wissenschaftlich sinnvollen Ct (Cutoff-Wert). Dieser oder ein anderer Cycler-Wert ist im eigentlichen Corman- Drosten-Papier nirgends zu finden.

4. Biomolekulare Validierungen
Um festzustellen, ob es sich bei den amplifizierten Produkten tatsächlich um SARS-CoV-2-Gene handelt, ist eine biomolekulare Validierung der amplifizierten PCR-Produkte unerlässlich. Für einen diagnostischen Test ist diese Validierung ein absolutes Muss.
Die Validierung der PCR-Produkte sollte entweder durch Ausführen des PCR-Produkts in einem 1%igen Agarose-EtBr-Gel zusammen mit einem Größenindikator (DNA-Lineal oder DNA-Leiter) erfolgen, damit die Größe des Produkts abgeschätzt werden kann. Die Größe muss mit der berechneten Größe des Amplifikationsprodukts übereinstimmen. Noch besser ist es jedoch, das Amplifikationsprodukt zu sequenzieren. Letzteres gibt 100%ige Sicherheit über die Identität des Amplifikationsprodukts. Ohne molekulare Validierung kann man sich der Identität der amplifizierten PCR-Produkte nicht sicher sein. In Anbetracht der oben beschriebenen schweren Designfehler können die amplifizierten PCR-Produkte alles Mögliche sein.
Ebenfalls nicht im Corman-Drosten Paper erwähnt ist der Fall von kleinen Fragmenten der qPCR (um 100bp): Es könnte sich um ein 1,5% Agarosegel oder sogar um ein Acrylamidgel handeln.
Die Tatsache, dass diese PCR-Produkte nicht auf molekularer Ebene validiert wurden, ist ein weiterer eklatanter Fehler des Protokolls, der jeden darauf basierenden Test als spezifisches diagnostisches Werkzeug zur Identifizierung des SARS-CoV-2-Virus unbrauchbar macht.
5. Positiv- und Negativkontrollen zur Bestätigung/Verweigerung des spezifischen Virusnachweises.
Die in der Arbeit von Corman-Drosten beschriebene unbestätigte Annahme ist, dass SARS-CoV-2 das einzige Virus aus der Gruppe der SARS-ähnlichen Beta-Coronaviren ist, das derzeit Infektionen beim Menschen verursacht. Die Sequenzen, auf denen ihre PCR-Methode basiert, sind in silico-Sequenzen, die von einem Labor in China zur Verfügung gestellt wurden [23], da den Autoren zum Zeitpunkt der Entwicklung des PCR-Tests kein Kontrollmaterial von infektiösem („live“) oder inaktiviertem SARS-CoV-2 zur Verfügung stand. Der PCR-Test wurde daher unter Verwendung der Sequenz des bekannten SARS-CoV als Kontrollmaterial für die Sarbeco-Komponente konzipiert (Dr. Meijer, Co-Autor Corman-Drosten paper in einem E-Mail-Austausch mit Dr. Peter Borger) [2].
Alle Personen, die mit dem RT-PCR-Test positiv getestet werden, wie in der Corman-Drosten-Arbeit beschrieben, werden als positiv für SARS-CoV-2-Infektionen angenommen. Es gibt drei schwerwiegende Fehler in ihrer Annahme. Erstens kann ein positiver Test auf die im Corman-Drosten-Paper beschriebenen RNA-Moleküle nicht mit einer „Infektion mit einem Virus“ gleichgesetzt werden. Ein positiver RT-PCR-Test zeigt lediglich das Vorhandensein von viralen RNA-Molekülen an. Wie unter Punkt 1d (oben) gezeigt, war der Corman-Drosten-Test nicht für den Nachweis des Virus in voller Länge ausgelegt, sondern nur für den Nachweis eines Fragments des Virus. Wir kamen bereits zu dem Schluss, dass dies den Test als diagnostischen Test für SARS-Virusinfektionen als ungeeignet einstuft.
Zweitens, und das ist von großer Bedeutung, wurde die Funktionalität des publizierten RT-PCR-Tests nicht unter Verwendung einer Positivkontrolle (isolierte SARS-CoV-2-RNA) nachgewiesen, die ein wesentlicher wissenschaftlicher Goldstandard ist.
Drittens heißt es im Corman-Drosten-Papier:
„Um zu zeigen, dass die Assays andere fledermausassoziierte SARS-verwandte Viren nachweisen können, haben wir den E-Gen-Assay verwendet, um sechs Fledermaus-Kotproben zu testen, die von Drexler et al. […] und Muth et al. […] zur Verfügung gestellt wurden. Diese virus-positiven Proben stammten von europäischen rhinolophiden Fledermäusen. Der Nachweis dieser phylogenetischen Ausreißer innerhalb der SARS-verwandten CoV-Klade deutet darauf hin, dass wahrscheinlich alle asiatischen Viren nachgewiesen werden können. Dies würde theoretisch eine breite Sensitivität auch bei mehrfachem, unabhängigem Erwerb von Virusvarianten aus einem tierischen Reservoir gewährleisten.“
Diese Aussage zeigt, dass das im RT-PCR-Test verwendete E-Gen, wie in der Arbeit von Corman-Drosten beschrieben, nicht spezifisch für SARS-CoV-2 ist.
Die E-Gen-Primer detektieren auch ein breites Spektrum von anderen SARS-Viren.
Das Genom des Coronavirus ist das größte aller RNA-Viren, die den Menschen infizieren, und sie haben alle eine sehr ähnliche molekulare Struktur. Dennoch haben SARS-CoV1 und SARS-CoV-2 zwei hochspezifische genetische Fingerabdrücke, die sie von den anderen Coronaviren unterscheiden. Erstens ist eine einzigartige Fingerprint-Sequenz (KTFPPTEPKKDKKKK) im N-Protein von SARS-CoV und SARS-CoV-2 vorhanden [13,14,15]. Zweitens enthalten sowohl SARS-CoV1 als auch SARS-CoV2 das HE-Protein nicht, während alle anderen Coronaviren dieses Gen besitzen [13, 14]. Um also ein SARS-CoV1- und SARS-CoV-2-PCR-Produkt spezifisch nachzuweisen, hätte die oben genannte Region im N-Gen als Amplifikationsziel gewählt werden müssen. Ein zuverlässiger diagnostischer Test sollte sich auf diese spezifische Region im N-Gen als Bestätigungstest konzentrieren. Die PCR für dieses N-Gen wurde in der Drosten-Corman-Arbeit nicht weiter validiert und auch nicht als Testgen empfohlen, weil sie mit der SARS-CoV-Originalsonde „nicht so empfindlich“ war [1].
Außerdem macht das Fehlen des HE-Gens sowohl bei SARS-CoV1 als auch bei SARS-CoV-2 dieses Gen zur idealen Negativkontrolle, um andere Coronaviren auszuschließen. Das Corman-Drosten-Papier enthält diese Negativkontrolle nicht und auch keine anderen Negativkontrollen. Der PCR-Test in der Arbeit von Corman-Drosten enthält also weder eine eindeutige Positivkontrolle noch eine Negativkontrolle zum Ausschluss anderer Coronaviren. Dies ist ein weiterer wesentlicher Konstruktionsfehler, der den Test als für die Diagnose ungeeignet einstuft.
6. Standardarbeitsanweisung (SOP) ist nicht verfügbar
Es sollte eine Standard Operational Procedure (SOP) zur Verfügung stehen, die die oben genannten Parameter eindeutig spezifiziert, so dass alle Laboratorien in der Lage sind, die identischen gleichen Testbedingungen einzurichten. Eine validierte, allgemeingültige SOP ist unerlässlich, da sie den Datenvergleich innerhalb und zwischen Ländern erleichtert. Es ist sehr wichtig, alle Primerparameter eindeutig zu spezifizieren. Wir stellen fest, dass dies nicht geschehen ist. Außerdem ist der Ct-Wert, der angibt, wann eine Probe als positiv oder negativ zu bewerten ist, nicht spezifiziert. Es ist auch nicht spezifiziert, wann eine Probe als mit SARS-CoV-Viren infiziert gilt. Wie oben gezeigt, kann der Test nicht zwischen Virus und Virusfragmenten unterscheiden, daher ist der Ct-Wert, der die Positivität anzeigt, von entscheidender Bedeutung. Dieser Ct-Wert hätte in der Standard Operational Procedure (SOP) spezifiziert und online gestellt werden müssen, damit alle Labore, die diesen Test durchführen, exakt die gleichen Randbedingungen haben. Es deutet auf mangelhafte Wissenschaft hin, dass eine solche SOP nicht existiert. Die Laboratorien sind somit frei, den Test so durchzuführen, wie sie es für richtig halten, was zu einer enormen Variationsbreite führt. Labore in ganz Europa stehen vor einer Vielzahl von Fragen: Welche Primer sollen bestellt werden? Welche Nukleotide sollen die undefinierten Stellen ausfüllen? Welchen Tm-Wert soll man wählen? Wie viele PCR-Zyklen sollen durchgeführt werden? Bei welchem Ct-Wert ist die Probe positiv? Und wann ist sie negativ? Und wie viele Gene sollen getestet werden? Sollen alle Gene getestet werden, oder nur das E- und das RpRd-Gen, wie in Tabelle 2 der Corman-Drosten-Arbeit gezeigt? Sollte auch das N-Gen getestet werden? Und was ist ihre Negativkontrolle? Was ist ihre Positivkontrolle?
Das Protokoll, so wie es beschrieben ist, ist leider sehr vage und so fehlerhaft aufgebaut, dass man in Dutzende von verschiedenen Richtungen gehen kann. Es scheint weder eine Standardisierung noch eine SOP zu geben, so dass nicht klar ist, wie dieser Test durchgeführt werden kann.
7. Folgen der unter 1-5 beschriebenen Fehler: falsch positive Ergebnisse.
Der in der Arbeit von Corman-Drosten beschriebene RT-PCR-Test enthält so viele molekularbiologische Designfehler (siehe 1-5), dass es nicht möglich ist, eindeutige Ergebnisse zu erhalten. Es ist unvermeidlich, dass dieser Test eine enorme Anzahl von so genannten „falsch-positiven“ Ergebnissen erzeugt. Die Definition von „falsch positiv“ ist eine Probe, die zunächst positiv ausfällt, aber nach einer erneuten Testung mit dem gleichen Test negativ ist. Falsch-Positive sind fälschlich positive Testergebnisse, also negative Proben, die positiv testen. Und genau das findet sich in der Corman-Drosten-Arbeit. Auf Seite 6 des Manuskript-PDFs zeigen die Autoren, dass selbst unter gut kontrollierten Laborbedingungen ein erheblicher Prozentsatz an falsch-positiven Ergebnissen mit diesem Test erzeugt wird:
„Bei vier einzelnen Testreaktionen wurde eine schwache anfängliche Reaktivität festgestellt, die jedoch bei einer erneuten Testung mit demselben Assay negativ war. Diese Signale wurden nicht mit einem bestimmten Virus in Verbindung gebracht, und für jedes Virus, bei dem eine anfängliche positive Reaktivität auftrat, gab es andere Proben, die das gleiche Virus in einer höheren Konzentration enthielten, aber nicht positiv getestet wurden. In Anbetracht der Ergebnisse der oben beschriebenen umfangreichen technischen Qualifizierung wurde der Schluss gezogen, dass diese anfängliche Reaktivität nicht auf die chemische Instabilität der Real-Time-PCR-Sonden zurückzuführen war und höchstwahrscheinlich auf Handhabungsprobleme, die durch die schnelle Einführung neuer diagnostischer Tests und Kontrollen während dieser Evaluierungsstudie verursacht wurden.“ [1]
Der erste Satz dieses Auszugs ist ein klarer Beleg dafür, dass der in der Corman-Drosten-Arbeit beschriebene PCR-Test falsch-positive Ergebnisse erzeugt. Selbst unter den gut kontrollierten Bedingungen des hochmodernen Charité-Labors sind 4 von 310 Primär-Tests per Definition falsch positiv. Vier negative Proben wurden zunächst positiv getestet, waren dann aber bei der Wiederholungsuntersuchung negativ. Dies ist das klassische Beispiel für ein falsches Positiv. In diesem Fall werden sie von den Autoren nicht als falsch positiv identifiziert, was intellektuell unredlich ist.
Eine weitere verräterische Beobachtung in dem obigen Auszug ist, dass die Autoren die falsch-positiven Ergebnisse mit „Handhabungsproblemen, die durch die schnelle Einführung neuer diagnostischer Tests verursacht werden“ erklären. Stellen Sie sich die Labore vor, die den Test einführen müssen, ohne alle notwendigen Informationen zu haben, die normalerweise in einer SOP beschrieben sind.
8. Das Corman-Drosten-Papier wurde nicht peer-reviewed
Vor der formalen Veröffentlichung in einer wissenschaftlichen Zeitschrift werden wissenschaftliche und medizinische Artikel traditionell durch ein „Peer-Review“ begutachtet. In diesem Prozess nehmen die Redakteure der Zeitschrift Ratschläge von verschiedenen Experten („Gutachtern“) an, die die Arbeit bewertet haben und möglicherweise Schwächen in den Annahmen, Methoden und Schlussfolgerungen identifizieren. Normalerweise veröffentlicht eine Zeitschrift einen Artikel erst dann, wenn die Redakteure überzeugt sind, dass die Autoren die Bedenken der Gutachter berücksichtigt haben und dass die präsentierten Daten die in der Arbeit gezogenen Schlussfolgerungen unterstützen.“ Dieser Prozess ist auch für Eurosurveillance beschrieben [16].
Das Corman-Drosten-Paper wurde am 21. Januar 2020 bei Eurosurveillance eingereicht und am 22. Januar 2020 zur Veröffentlichung angenommen. Am 23. Januar 2020 war das Papier online. Am 13. Januar 2020 wurde die Version 1-0 des Protokolls auf der offiziellen WHO-Website veröffentlicht [17], aktualisiert am 17. Januar 2020 als Dokumentversion 2-1 [18], noch bevor das Corman-Drosten-Paper am 23. Januar bei Eurosurveillance veröffentlicht wurde.
Normalerweise ist ein Peer-Review ein zeitaufwändiger Prozess, da mindestens zwei Experten aus dem Fachgebiet das eingereichte Papier kritisch lesen und kommentieren müssen. Unserer Meinung nach wurde diese Arbeit nicht von einem Peer-Review-Verfahren geprüft. Vierundzwanzig Stunden sind einfach nicht genug, um ein gründliches Peer-Review durchzuführen. Unsere Schlussfolgerung wird durch die Tatsache gestützt, dass wir eine enorme Anzahl von sehr schwerwiegenden Konstruktionsfehlern gefunden haben, die den PCR-Test als diagnostisches Werkzeug zur Identifizierung des SARS-CoV-2-Virus völlig ungeeignet machen. Jeder Molekularbiologe, der mit dem RT-PCR-Design vertraut ist, hätte die schwerwiegenden Fehler im Corman-Drosten-Papier schon vor dem eigentlichen Review-Prozess leicht erkennen können. Wir haben Eurosurveillance am 26. Oktober 2020 gebeten, uns eine Kopie des Peer-Review-Berichts zu schicken. Bis heute haben wir diesen Bericht nicht erhalten, und in einem Schreiben vom 18. November 2020 lehnte das ECDC als Gastgeber für Eurosurveillance den Zugang ab, ohne substanzielle wissenschaftliche Gründe für ihre Entscheidung zu liefern. Im Gegenteil, sie schreiben, dass „die Offenlegung den Zweck wissenschaftlicher Untersuchungen untergraben würde.“ [24].
9. Autoren als Redakteure
Ein letzter Punkt ist von großer Bedeutung. Es stellt sich heraus, dass zwei Autoren des Corman-Drosten-Papiers, Christian Drosten und Chantal Reusken, auch Mitglieder des Redaktionsausschusses dieser Zeitschrift sind [19]. Es besteht also ein schwerer Interessenkonflikt, der den Verdacht erhärtet, dass das Papier nicht peer-reviewed wurde. Es hat den Anschein, dass die schnelle Veröffentlichung nur deshalb möglich war, weil die Autoren auch dem Redaktionsbeirat von Eurosurveillance angehörten. Diese Praxis wird als Gefährdung der wissenschaftlichen Integrität eingestuft.
ZUSAMMENFASSENDER KATALOG DER IN DEM PAPIER GEFUNDENEN FEHLER
Das Corman-Drosten-Papier enthält die folgenden spezifischen Fehler:
- Es gibt keinen spezifizierten Grund für die Verwendung dieser extrem hohen Konzentrationen von Primern in diesem Protokoll. Die beschriebenen Konzentrationen führen zu erhöhten unspezifischen Bindungen und PCR-Produktamplifikationen, was den Test als spezifisches Diagnoseinstrument zur Identifizierung des SARS-CoV-2-Virus ungeeignet macht.
- Sechs nicht spezifizierte wackelige Positionen führen zu einer enormen Variabilität in den realen Laborimplementierungen dieses Tests; die verwirrende unspezifische Beschreibung im Corman-Drosten-Papier ist nicht als Standard-Arbeitsprotokoll geeignet, was den Test als spezifisches Diagnoseinstrument zur Identifizierung des SARS-CoV-2-Virus ungeeignet macht.
- Der Test kann nicht zwischen dem ganzen Virus und viralen Fragmenten unterscheiden. Daher kann der Test nicht als Diagnostikum für intakte (infektiöse) Viren verwendet werden, was den Test als spezifisches Diagnoseinstrument zur Identifizierung des SARS-CoV-2-Virus ungeeignet macht und Rückschlüsse auf das Vorliegen einer Infektion zulässt.
- Eine Differenz von 10° C in Bezug auf die Annealing-Temperatur Tm für Primerpaar1 (RdRp_SARSr_F und RdRp_SARSr_R) macht den Test ebenfalls ungeeignet als spezifisches Diagnoseinstrument zur Identifizierung des SARS-CoV-2-Virus.
- Ein schwerwiegender Fehler ist das Fehlen eines Ct-Wertes, bei dem eine Probe als positiv und negativ angesehen wird. Dieser Ct-Wert findet sich auch nicht in den Folgeanträgen, was den Test als spezifisches Diagnosewerkzeug zum Nachweis des SARS-CoV-2-Virus ungeeignet macht.
- Die PCR-Produkte wurden nicht auf molekularer Ebene validiert. Diese Tatsache macht das Protokoll als spezifisches Diagnosewerkzeug zum Nachweis des SARS-CoV-2-Virus unbrauchbar.
- Der PCR-Test enthält weder eine eindeutige Positivkontrolle, um seine Spezifität für SARS-CoV-2 zu bewerten, noch eine Negativkontrolle, um das Vorhandensein anderer Coronaviren auszuschließen, wodurch der Test als spezifisches Diagnoseinstrument zur Identifizierung des SARS-CoV-2-Virus ungeeignet ist.
- Das Testdesign im Corman-Drosten-Papier ist so vage und fehlerhaft, dass man in Dutzende von verschiedenen Richtungen gehen kann; nichts ist standardisiert und es gibt keine SOP. Dies stellt die wissenschaftliche Validität des Tests stark in Frage und macht ihn als spezifisches Diagnoseinstrument zur Identifizierung des SARS-CoV-2-Virus ungeeignet.
- Höchstwahrscheinlich wurde das Corman-Drosten-Papier nicht peer-reviewed, was den Test als spezifisches Diagnoseinstrument zur Identifizierung des SARS-CoV-2-Virus ungeeignet macht.
- Wir finden schwerwiegende Interessenkonflikte bei mindestens vier Autoren, zusätzlich zu der Tatsache, dass zwei der Autoren des Corman-Drosten Papers (Christian Drosten und Chantal Reusken) Mitglieder des Redaktionsausschusses von Eurosurveillance sind. Am 29. Juli 2020 wurde ein Interessenkonflikt hinzugefügt (Olfert Landt ist Geschäftsführer von TIB-Molbiol; Marco Kaiser ist Senior Researcher bei GenExpress und dient als wissenschaftlicher Berater für TIB-Molbiol), der in der ursprünglichen Version nicht deklariert war (und in der PubMed-Version immer noch fehlt); TIB-Molbiol ist die Firma, die „als erste“ PCR-Kits (Light Mix) auf der Basis des im Corman-Drosten-Manuskript veröffentlichten Protokolls herstellte, und nach eigenen Worten diese PCR-Test-Kits schon vor der Publikation vertrieben hat [20]; außerdem haben Victor Corman & Christian Drosten ihre zweite Zugehörigkeit nicht erwähnt: das kommerzielle Testlabor „Labor Berlin“. Beide sind dort für die Virendiagnostik verantwortlich [21] und die Firma ist im Bereich der Real-Time-PCR-Tests tätig.
Im Rahmen unserer erneuten Überprüfung des im Corman-Drosten-Paper beschriebenen Testprotokolls zum Nachweis von SARS-CoV-2 haben wir entsprechende Fehler und inhärente Irrtümer festgestellt, die den SARS-CoV-2-PCR-Test unbrauchbar machen.
SCHLUSSFOLGERUNG
Die Entscheidung, welche Testprotokolle veröffentlicht und allgemein zugänglich gemacht werden, liegt in den Händen von Eurosurveillance. Eine Entscheidung, die offensichtlichen Fehler im Corman-Drosten-Papier anzuerkennen, hat den Vorteil, dass die Kosten und das Leiden der Menschen in Zukunft stark minimiert werden.
Ist es nicht im besten Interesse von Eurosurveillance, dieses Papier zurückzuziehen? Unsere Schlussfolgerung ist klar. Angesichts all der enormen PCR-Protokoll-Designmängel und Fehler, die hier beschrieben werden, kommen wir zu dem Schluss: Im Rahmen der wissenschaftlichen Integrität und Verantwortung bleibt keine große Wahl.
REFERENZEN
[1] Corman Victor M, Landt Olfert, Kaiser Marco, Molenkamp Richard, Meijer Adam, Chu Daniel KW, Bleicker Tobias, Brünink Sebastian, Schneider Julia, Schmidt Marie Luisa, Mulders Daphne GJC, Haagmans Bart L, van der Veer Bas, van den Brink Sharon, Wijsman Lisa, Goderski Gabriel, Romette Jean-Louis, Ellis Joanna, Zambon Maria, Peiris Malik, Goossens Herman, Reusken Chantal, Koopmans Marion PG, Drosten Christian. Nachweis des neuartigen Coronavirus 2019 (2019-nCoV) durch Echtzeit-RT-PCR. Euro Surveill. 2020;25(3):pii=2000045. https://doi.org/10.2807/1560-7917.ES.2020.25.3.2000045
[2] E-Mail-Kommunikation zwischen Dr. Peter Borger & Dr. Adam Meijer: Supplementary Material
[3] Jafaar et al., Correlation Between 3790 Quantitative Polymerase Chain Reaction-Positives Samples and Positive Cell Cultures, Including 1941 Severe Acute Respiratory Syndrome Coronavirus 2 Isolates. https://academic.oup.com/cid/advance-article/doi/10.1093/cid/ciaa1491/5912603
[4] BBC, 21. Januar 2020: https://www.bbc.com/news/world-asia-china-51185836;
Archiv: https://archive.is/0qRmZ
[5] Google Analytics – COVID19-Todesfälle weltweit: https://bit.ly/3fndemJ
Archiv: https://archive.is/PpqEE
[6] Laboruntersuchung für COVID-19 Emergency Response Technical Centre, NIVD unter
China CDC 15. März 2020: http://www.chinacdc.cn/en/COVID19/202003/P020200323390321297894.pdf
[7] Real-Time PCR Handbook Life Technologies: https://www.thermofisher.com/content/dam/LifeTech/global/Forms/PDF/real-time-pcr-handbuch.pdf
Nolan T, Huggett J, Sanchez E.Good practice guide for the application of quantitative PCR (qPCR) First Edition 2013
[8] Trestan Pillonel et al, Letter to the editor: SARS-CoV-2 detection by real-time RT-PCR: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7268274/
[9] Kurkela, Satu, und David WG Brown. „Molekular-diagnostische Techniken“. Medicine 38.10
(2009): 535-540.
[10] Wolfel et al. „Virologische Beurteilung von hospitalisierten Patienten mit COVID-2019“.
https://www.nature.com/articles/s41586-020-2196-x
[11] Thermofischer Primer Dimer Web Tool: https://www.thermofisher.com/us/en/home/brands/thermo-scientific/molecular-biology/molecular-biology-learning-center/molecular-biology-resource-library/thermo-scientific-web-tools/multiple-primer-analyzer.html
Ergänzendes Material
[12] Primer-BLAST, NCBI – National Center for Biotechnology Information: https://www.ncbi.nlm.nih.gov/tools/primer-blast/
[13] Marra MA, Steven JMJ, Caroline RA, Robert AH, Angela BW et al. (2003) Science. Die
Genomsequenz des SARS-assoziierten Coronavirus. Science 300(5624): 1399-1404.
14] Schweres Akutes Respiratorisches Syndrom Coronavirus 2 Isolat Wuhan-Hu-1, vollständiges
Genom: https://www.ncbi.nlm.nih.gov/nuccore/MN908947
[15] Borger P. Ein SARS-ähnliches Coronavirus wurde erwartet, aber es wurde nichts getan, um vorbereitet zu sein. Am J Biomed Sci Res 2020. https://biomedgrid.com/pdf/AJBSR.MS.ID.001312.pdf
https://www.researchgate.net/publication/341120750_A_SARS-like_Coronavirus_was_Expected_but_nothing_was_done_to_be_Prepared;
Archiv: https://archive.is/i76Hu
[16] Eurosurveillance paper evaluation / review process: https://www.eurosurveillance.org/evaluation
[17] Offizielle Empfehlung des Corman-Drosten-Protokolls & Manuskripts durch die WHO, veröffentlicht am 13. Januar 2020 als Version 1.0 des Dokuments:
https://www.who.int/docs/default-source/coronaviruse/wuhan-virus-assay-v1991527e5122341d99287a1b17c111902.pdf;
archive: https://bit.ly/3m3jXVH
18] Offizielle WHO-Empfehlung für das Corman / Drosten RT-qPCR-Protokoll, das direkt aus der Eurosurveillance-Publikation abgeleitet ist, Dokument-Version 2-1, veröffentlicht am 17. Januar 2020: https://www.who.int/docs/default-source/coronaviruse/protocol-v2-1.pdf?sfvrsn=a9ef618c_2
[19] Eurosurveillance-Redaktion, 2020: https://www.eurosurveillance.org/upload/site-assets/imgs/2020-09-Editorial%20Board%20PDF.pdf;
Archiv: https://bit.ly/2TqXBjX
[20] Gebrauchsanweisung LightMix SarbecoV E-gene plus EAV Control, TIB-Molbiol & Roche Molecular Solutions, 11. Januar 2020:
https://www.roche-as.es/lm_pdf/MDx_40-0776_96_Sarbeco-E-gene_V200204_09164154001_(1).pdf
Archiv, Zeitstempel – 11. Januar 2020: https://archive.is/Vulo5;
Archiv: https://bit.ly/3fm9bXH
[21] Christian Drosten & Victor Corman, verantwortlich für die virale Diagnostik im Labor Berlin:
https://www.laborberlin.com/fachbereiche/virologie/
Archiv: https://archive.is/CDEUG
[22] Tom Jefferson, Elizabeth Spencer, Jon Brassey, Carl Heneghan Virenkulturen zur COVID-19 Infektiositätsbestimmung. Systematische Übersichtsarbeit. Systematische Übersichtsarbeit doi:
https://doi.org/10.1101/2020.08.04.20167932
https://www.medrxiv.org/content/10.1101/2020.08.04.20167932v4
[23] Kim et al.,The Architecture of SARS-CoV-2 Transcriptome:
https://www.sciencedirect.com/science/article/pii/S0092867420304062
[24] ECDC-Antwort an Dr. Peter Borger, 18. November 2020:
Ergänzendes Material
[25] Prof. Dr. Ulrike Kämmerer & Team, Übersicht & Primer-BLAST-Tabelle:
Ergänzendes Material
Zusätzliche Literatur:
Beschreibung RT-PCR RKI Deutschland, auf Seite 10 dieses Links:
https://www.rki.de/DE/Content/Gesundheitsmonitoring/Gesundheitsberichterstattung/GBEDownloadsJ/JoHM_S5_2020_Studienprotokoll_CORONA_MONITORING_lokal.pdf?__blob=publicationFile
Zugehörigkeit der Autoren:
1) Dr. Pieter Borger (MSc, PhD), Molekulargenetiker, W+W Research Associate, Lörrach, Deutschland
2) Rajesh Kumar Malhotra (Artist Alias: Bobby Rajesh Malhotra), Ehemaliger 3D Artist / Wissenschaftliche Visualisierungen am CeMM – Zentrum für Molekulare Medizin der Österreichischen Akademie der Wissenschaften (2019-2020), Universität für angewandte Kunst – Abteilung für Digitale Kunst Wien, Österreich
3) Dr. Michael Yeadon BSs(Hons) Biochem Tox U Surrey, PhD Pharmakologie U Surrey. Geschäftsführer, Yeadon Consulting Ltd, ehemaliger Pfizer Chief Scientist, Großbritannien
4) Dr. Clare Craig MA, (Cantab) BM, BCh (Oxon), FRCPath, Vereinigtes Königreich
5) Kevin McKernan, BS Emory University, Chief Scientific Officer, Gründer von Medical Genomics, hat die Sequenzierungspipeline am WIBR/MIT für das Human Genome Project entwickelt, erfand und entwickelte den SOLiD-Sequenzer, erhielt Patente in Bezug auf PCR, DNA-Isolierung und Sequenzierung, USA
6) Prof. Dr. Klaus Steger, Klinik für Urologie, Kinderurologie und Andrologie, Molekulare Andrologie, Biomedizinisches Forschungszentrum der Justus-Liebig-Universität, Gießen, Deutschland
7) Dr. Paul McSheehy (BSc, PhD), Biochemiker & Industrie-Pharmakologe, Lörrach, Deutschland
8) Dr. Lidiya Angelova, MSc in Biologie, PhD in Mikrobiologie, ehemalige Forscherin am National Institute of Allergy and Infectious Diseases (NIAID), Maryland, USA
9) Dr. Fabio Franchi, Ehemaliger Dirigente Medico (M.D) in einer Infektionsabteilung, spezialisiert auf „Infektionskrankheiten“ und „Hygiene und Präventivmedizin“, Società Scientifica per il Principio di Precauzione (SSPP), Italien
10) Dr. med. Thomas Binder, Internist und Kardiologe (FMH), Schweiz
11) Prof. Dr. med. Henrik Ullrich, Facharzt für Diagnostische Radiologie, Chefarzt am Zentrum für Radiologie des Collm Oschatz-Krankenhauses, Deutschland
12) Prof. Dr. Makoto Ohashi, Professor emeritus, PhD in Mikrobiologie und Immunologie, Tokushima Universität, Japan
13) Dr. Stefano Scoglio, B.Sc. Ph.D., Mikrobiologe, Ernährungswissenschaftler, Italien
14) Dr. Marjolein Doesburg-van Kleffens (MSc, PhD), Fachärztin für Labormedizin (klinische Chemie), Maasziekenhuis Pantein, Beugen, Niederlande
15) Dr. Dorothea Gilbert (MSc, PhD), PhD Umweltchemie und Toxikologie. DGI Beratungsdienste, Oslo, Norwegen
16) Dr. Rainer J. Klement, PhD. Abteilung für Strahlenonkologie, Leopoldina Krankenhaus Schweinfurt, Deutschland
17) Dr. Ruth Schruefer, PhD, Humangenetik/Immunologie, München, Deutschland,
18) Dra. Berber W. Pieksma, Allgemeinmediziner, Niederlande
19) Dr. med. Jan Bonte (GJ), Beratender Neurologe, Die Niederlande
20) Dr. Bruno H. Dalle Carbonare (Molekularbiologe), IP-Spezialist, BDC Basel, Schweiz
21) Dr. Kevin P. Corbett, MSc Krankenpflege (Kings College London) PhD (London South Bank) Sozialwissenschaften (Science & Technology Studies) London, England, Vereinigtes Königreich
22) Prof. Dr. Ulrike Kämmerer, Fachärztin für Virologie / Immunologie / Humanbiologie / Zellbiologie, Universitätsklinikum Würzburg, Deutschland
Beiträge der Autoren:
PB: Planung und Durchführung der Analysen und Forschung, Konzeption des Manuskripts.
BRM: Planung und Durchführung der Forschung, Konzeption der Abbildungen und des Manuskripts.
MY: Korrekturlesen der Analysen und Recherchen.
KMcK: Durchführung der Analysen und Recherchen, Konzeption des Manuskripts.
KS: Führte die Analysen und Recherchen durch.
PMcS: Lektorat der Analysen und Recherchen.
LA: Lektorat der Analysen und Recherchen.
FF: Lektorat der Analysen und Recherchen.
TB: Lektorat der Analysen und Recherchen.
HU: Lektorat der Analysen und Recherchen.
MO: Lektorat der Analysen und Recherchen.
SS: Lektorat der Analysen und Recherchen.
MDvK: Lektorat der Analysen und Recherchen.
DG: Lektorat der Analysen und Recherchen.
RJK: Lektorat der Analysen und Recherchen.
RS: Korrekturlesen der Analysen und Recherchen, sowie des Manuskripts.
BWK: Lektorat der Analysen und Recherchen.
RvV: Lektorat der Analysen und Recherchen.
JB: Lektorat der Analysen und Recherchen.
KC: Lektorat der Analysen und Recherchen.
UK: Plante und führte die Analysen und Recherchen durch und konzipierte das Manuskript.
Zusätzliche Korrekturleser:
Saji N Hameed, Umweltinformatik, Universität von Aizu, Tsuruga, Ikki-machi, Aizuwakamatsu-shi, Fukushima, Japan
Howard R. Steen, MA Chem. Eng. Cantab, Ehemaliger Forschungsleiter, Deutschland
Nachtrag
Aktualisierung 2.12.2020:
Autorenbeitrag Dr. Michael Yeadon geändert in:
Korrekturlesen der Analysen und Recherche.
Autorenzugehörigkeit Kevin Mckernan geändert in:
Medizinische Genomik.
229

Keine Kommentare möglich.